Webseite "Aquaria" liefert Forschern 3D-Strukturen von Proteinen
Virtuelle Reise ins Innere der Proteine
Eiweiße, auch Proteine genannt, übernehmen in unserem Körper essentielle Aufgaben. Bekannte Proteine sind zum Beispiel Insulin, das den Blutzuckerspiegel reguliert und Hämoglobin, das für den Sauerstofftransport im Blut verantwortlich ist. Auch Antikörper zur Abwehr von Krankheitserregern sind Proteine. Die dreidimensionale Struktur der Eiweiße ist dabei entscheidend für ihre Funktion.
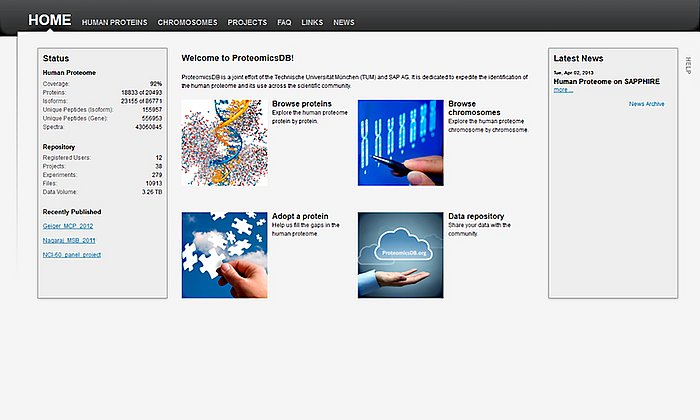
Um Experimente zu planen oder Antikörper herzustellen, ist es für Wissenschaftler essentiell, so viele Informationen über die Proteinen zur Verfügung zu haben wie möglich. Zwar sind viele Daten vorhanden. "Die Suche nach Proteinstrukturen war allerdings bisher mühsam und erforderte Expertenwissen", erklärt Andrea Schafferhans vom Lehrstuhl für Bioinformatik der TUM. Gemeinsam mit Dr. Seán O’Donoghue vom Garvan Institute of Medical Research und CSIRO entwickelte sie daher die Datenbank "Aquaria". "In Aquaria sind bereits alle Daten aufbereitet", sagt Schafferhans.
Ihre Ergebnisse publizierten die Wissenschaftler im Fachmagazin "Nature Methods". An dem Projekt, das 2009 startete, war ein internationales Team von etwa einem Dutzend Programmierern und Bioinformatikern beteiligt.
46 Millionen Computermodelle
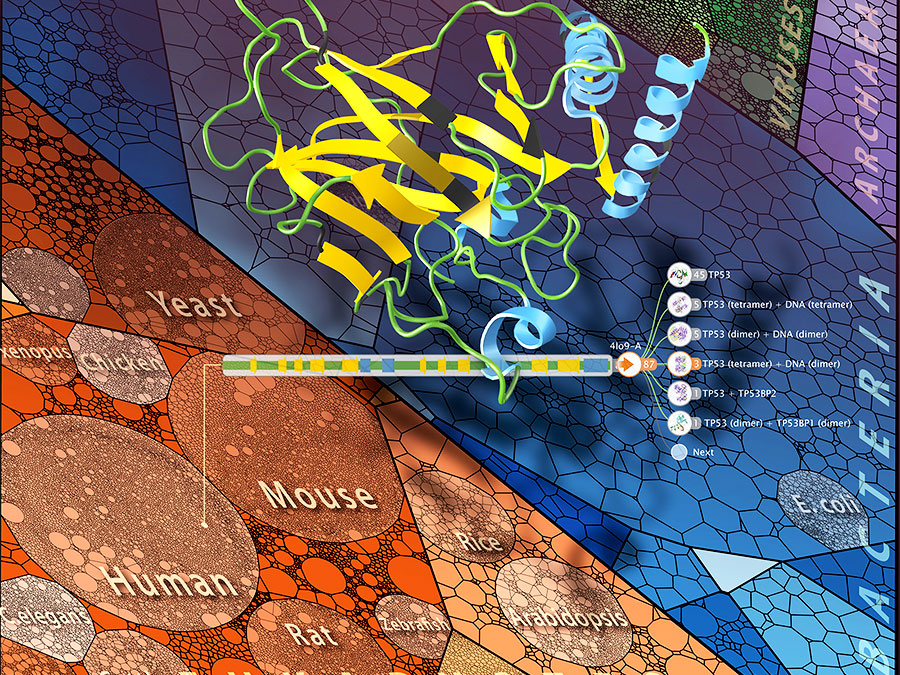
"Aquaria" errechnet die Struktur der meisten Eiweiße, basierend auf bereits bekannten Datensätzen. "Nur für fünf Prozent der ausführlich beschriebenen Proteine ist deren Struktur experimentell eindeutig geklärt", sagt Schafferhans. Das Prinzip hinter der Berechnung: Proteine bestehen aus Aminosäuren. Die Abfolge, also Sequenz dieser Bausteine ist für viele Eiweiße bekannt. Die genaue dreidimensionale Struktur jedoch nur für wenige. Die Wissenschaftler nahmen über 500.000 Proteinsequenzen in die Datenbank auf und verglichen diese mit den 100.000 bekannten Eiweißstrukturen aus der Proteinstruktur-Datenbank PDB. Daraus ergaben sich etwa 46 Millionen Computermodelle.
Forscher müssen auf der öffentlich zugänglichen Webseite (http://aquaria.ws) nur den Namen des gesuchten Proteins in die Suchmaske eingeben. Das Programm lädt eine Seite, die ein Modell der dreidimensionalen Struktur des Eiweißes anzeigt. Ist noch keine experimentell bestimmte Struktur vorhanden, wird die wahrscheinlichste Variante errechnet.
Es können aber noch viele zusätzliche Informationen abgerufen werden. Wissenschaftler können zum Beispiel erkunden, wie Veränderungen einzelner Aminosäuren die Struktur des Eiweißes verändern – und damit dessen Funktion im Körper. "Eine krankheitsauslösende Sequenzvariante kann so in ihrem Umfeld gezeigt werden", erklärt Prof. Burkhard Rost. "Diese Einsicht kann für die Entwicklung eines Medikaments entscheidend sein."
Für die Zukunft sollen noch weitere Funktionen in "Aquaria" ergänzt werden, sagt Schafferhans. "Wir planen unter anderem, Wechselwirkungen mit kleinen Molekülen wie Hormonen und Medikamenten genauer zu untersuchen und durchsuchbar zu machen."
Dieses Projekt entstand im Rahmen der Humboldt-Professor von Burkhard Rost und wurde mit Mitteln der Humboldtstiftung gefördert. Das Hosting von "Aquaria" wurde von Amazon AWS unterstützt.
Publikation:
Seán I O'Donoghue, Kenneth S Sabir, Maria Kalemanov, Christian Stolte, Benjamin Wellmann, Vivian Ho, Manfred Roos, Nelson Perdigão, Fabian A Buske, Julian Heinrich, Burkhard Rost & Andrea Schafferhans.
Aquaria: simplifying discovery and insight from protein structures, Nature Methods 12, 98–99 (2015), doi: 10.1038/nmeth.3258
Kontakt:
Andrea Schafferhans
Technische Universität München
I12, Chair of Prof. Rost
Tel.: +49 289 17833
andrea.schafferhans@rostlab.org
Weitere Informationen:
Anleitung zur Nutzung der Datenbank Aquaria auf Youtube (englisch)
Arbeitsgruppe Prof. Burkhard Rost
Technische Universität München
Corporate Communications Center
- Stefanie Reiffert
- reiffert@zv.tum.de
- presse@tum.de
- Teamwebsite