Enzymbehandlung von Hautproben verbessert die Mikrobiom-Analyse
Haut-Bakterien im Visier

Bei vielen Hautkrankheiten, beispielsweise Neurodermitis oder Akne, ist der bakterielle Schutzfilm der Haut geschädigt. „Unser Ziel ist es herauszufinden, welche Rolle die verschiedenen Hautbakterien bei solchen Erkrankungen spielen“, erklärt Dr. Martin Köberle, Leiter des Labors Dermatoinfektiologie am Klinikum Rechts der Isar der Technischen Universität München (TUM).
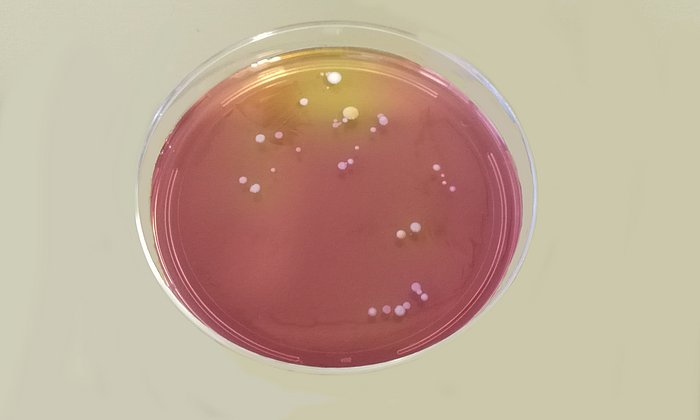
Bisher stießen Dermatologinnen und Dermatologen schnell an Grenzen, wenn sie sie die genaue Zusammensetzung des Mikrobioms untersuchen wollten. Der Grund: In der klassischen Kultur auf Agarplatten vermehren sich nicht alle Bakterien aus einem Hautabstrich gleich gut und gleich schnell – einzelne können daher übersehen werden. Neuere genetische Analyseverfahren haben den Nachteil, dass große Mengen an DNA-Sequenzen aus Hautzellen sowie Bruchstücke toter Bakterien in das Ergebnis mit einfließen, was die Aussagekraft mindert.
Genetische Spreu vom Weizen trennen
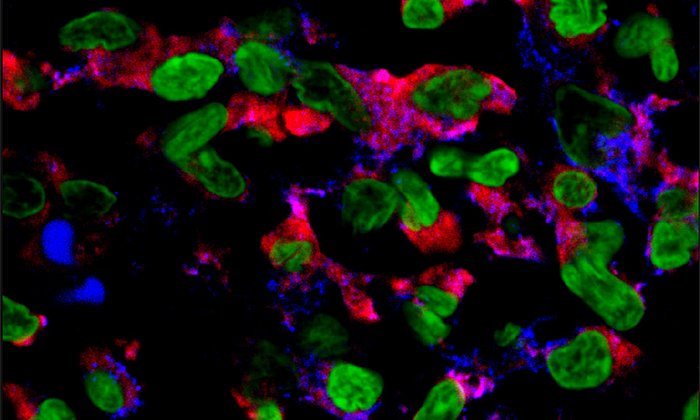
Köberle und der Biologe Dr. Yacine Amar, beide Mitarbeiter von Prof. Biedermann an der Klinik und Poliklinik für Dermatologie und Allergologie der TUM, haben zusammen mit einem internationalen und interdisziplinären Team eine Methode entwickelt, mit der sich die störende Fremd-DNA entfernen lässt: Die Forschenden nutzen dabei eine besondere Fähigkeit des Enzyms Benzonase. Dieses zerstört die Nukleotid-Ketten, die bei allen Lebewesen die Erbinformation tragen, indem sie diese in kleine Stücke zerlegen. Ausgenommen von dieser enzymatischen Zerstörung sind nur lebende Bakterien, deren Erbgut durch eine Zellwand geschützt ist.
Benzonase wird schon seit längerem eingesetzt, um beispielsweise Proteine aufzureinigen: Die Enzyme zerlegen alle störenden DNA- und RNA-Bruchstücke, diese können anschließend in einer Zentrifuge entfernt werden. Übrig bleiben die Proteine. Die Selektion der Hautbakterien funktioniert nach demselben Prinzip: Genstücke aus Hautzellen oder toten Bakterien werden vom Enzym zerkleinert und lassen sich abtrennen. Die übrig gebliebenen Bakterien kann man mechanisch zerstören und dann gezielt deren DNA untersuchen.
„Unsere Experimente haben gezeigt, dass sich mit dieser Methode tatsächlich die Fremd-DNA vollständig eliminieren und das Mikrobiom der Haut selektieren lässt“, sagt Projektleiter Yacine Amar. Im Labor hat er zunächst künstlich hergestellte Proben, bei denen menschliche Zellen, tote und lebende Bakterien nach einem strengen Protokoll gemischt worden waren, mit Benzonase vorbehandelt. „Das anschließend verwendete Verfahren, das sogenannte 16S Sequencing, hat sehr präzise die Zusammensetzung der intakten Bakterien widergegeben“, betont der Forscher. Auch die Analyse von echten Hautabstrichen verlief erfolgreich: In den Proben wurde keine Rest-DNA von toten Bakterien gefunden.
Eine wichtige Rolle wird diese Herangehensweise auch künftig in der Forschung spielen, davon ist Köberle überzeugt: „Die enzymbasierte Selektion von lebendigen Hautbakterien kann uns helfen, einerseits mikrobielle Biomarker für bestimmte dermatologische Krankheiten zu finden, andererseits aber auch die Bakterien zu identifizieren, die den Krankheitsverlauf positiv beeinflussen. Sie könnten vielleicht eines Tages gezielt für eine Therapie eingesetzt werden.“ Die neue Methode zur Mikrobiom-Analyse wird an der Klinik und Poliklinik für Dermatologie der TUM bereits in zahlreichen Kohortenstudien zu Hautkrankheiten angewandt.
Yacine Amar, Ilias Lagkouvardos, Rafaela L. Silva, Oluwaseun Ayodeji Ishola, Bärbel U. Foesel, Susanne Kublik, Anne Schöler, Sebastian Niedermeier, Rachela Bleuel, Alexander Zink, Klaus Neuhaus, Michael Schloter, Tilo Biedermann, Martin Köberle
Pre-digest of unprotected DNA by Benzonase improves the representation of living skin bacteria and efficiently depletes host DNA
Microbiome, 26. Mai 2021 9:123
DOI: 10.1186/s40168-021-01067-0
An den Untersuchungen beteiligt war neben der TUM auch das Helmholtz Zentrum München. Die Forschungsarbeit wurde unterstützt durch die Deutsche Forschungsgemeinschaft (DFG), das Bundesministerium für Bildung und Forschung (BMBF) sowie das Bundesministerium für Wirtschaft und Energie (BMWi). Förderung erteilte ferner der Luxembourg National Research Fund (FNR) sowie die Helmholtz-Gemeinschaft.
Technische Universität München
Corporate Communications Center
- Lisa Pietrzyk
- lisa.pietrzyk@tum.de
- presse@tum.de
- Teamwebsite
Kontakte zum Artikel:
Martin Köberle
Klinikum Rechts der Isar
der Technischen Universität München
Klinik und Poliklinik für Dermatologie und Allergologie
Biedersteiner Str. 29
D-80802 München
Tel.: 089/4140 - 3501
martin.koeberle@tum.de