Von genetischen Variationen über bindende Proteine zur Regulation von Genen
Proteomik hilft den Einfluss genetischer Variationen zu verstehen
Das Risiko für zahlreiche komplexe Krankheiten wie Typ-2-Diabetes wird durch Veränderungen an unserem Erbgut erhöht, der DNA. Ein Mitspieler bei der Krankheitsentstehung sind Einzelnukleotid-Polymorphismen, die Wissenschaftler nur kurz „Snips“ (SNPs - Single Nucleotide Polymorphisms) nennen. Bei SNPs treten Variationen eines einzelnen Basenpaares in einem DNA-Molekül auf, wobei die dadurch beeinflussten Mechanismen meist unklar sind.
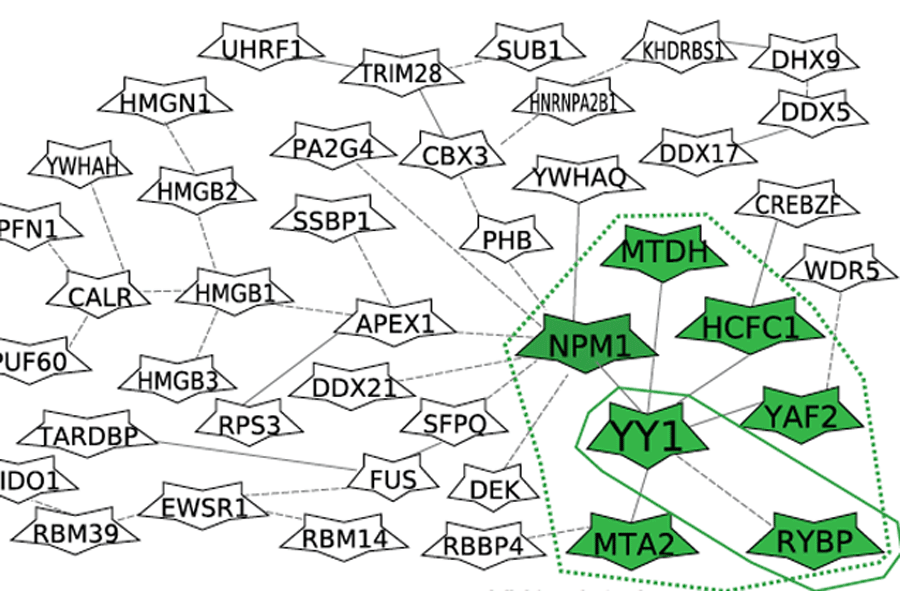
Es ist bekannt, dass manche dieser Risiko-SNPs die Regulation von Genen beeinflussen. Außerdem ist bekannt, dass die Genregulation von besonderen Proteinen gesteuert wird. Risiko-SNPs beeinflussen vermutlich, wie diese Proteine oder Proteinkomplexe an die DNA binden. „Bisher ist es Wissenschaftlern nur selten gelungen, bei krankheitsspezifischen SNPs eine unterschiedliche Bindung von genregulatorischen Protein Komplexen nachzuweisen“, erklärt Dr. Helmut Laumen von der Klinischen Kooperationsgruppe (KKG) Interaktion von Ernährung und Genetik bei Typ 2 Diabetes mellitus und der Pädiatrischen Ernährungsmedizin der TUM. Die KKG ist eine Kooperation des Helmholtz Zentrums München (Institut für Epidemiologie 2) und der Technischen Universität München (Else Kröner-Fresenius-Zentrum für Ernährungsmedizin).
„Interessant wäre insbesondere, mehr über die verschiedenen beteiligten Proteine zu erfahren, die einen starken Einfluss auf die Regulation von Genen haben“, sagt Laumen. Ein Team des Helmholtz-Zentrums München und der TUM hat diese durch SNP beeinflussten Mechanismen genauer analysiert und darüber eine Studie in „Nucleic Acids Research“ veröffentlicht. Das Team hat hochempfindliche massenspektrometrische Methoden eingesetzt, um damit bislang unbekannte Proteine und Proteinkomplexe zu identifizieren, deren Bindung an die DNA durch SNPs beeinflusst wird.
Lücke zwischen genetischen Variationen und dem Verständnis ihrer Funktion geschlossen
„Die bislang bestehende Lücke zwischen genetischen Variationen und dem Verständnis ihrer funktionellen Auswirkung auf den Organismus konnten wir schließen“, erzählt Dr. Stefanie Hauck, Leiterin der Abteilung Proteinanalytik und Core Facility Proteomics am Helmholtz Zentrum München.
„Konkret wenden wir diese Methode auf SNPs an, die für Typ-2-Diabetes und die Altersbedinge Makuladegeneration (AMD) relevant sind“, sagt Hauck. „Mit dem Verfahren lassen sich künftig für alle krankheitsassoziierten SNPs funktionell relevante Proteine identifizieren, um zu klären, welche Mechanismen hinter einem Krankheitsbild stecken.“ Das exakte Verständnis der molekularen Mechanismen, die durch regulatorische SNPs verändert werden, kann zur weiteren Entwicklung der personalisierten Medizin beitragen.
Weitere Informationen
An der Arbeit waren die Abteilung Proteinanalytik und Core Facility Proteomics (Helmholtz Zentrum München), die Klinische Kooperationsgruppe Klinischen Kooperationsgruppe Interaktion von Ernährung und Genetik bei Typ 2-Diabetes mellitus (Helmholtz Zentrum München, Technische Universität Mün-chen) und das Else Kröner-Fresenius-Zentrum für Ernährungsmedizin (Technische Universität München, Klinische Ernährungsmedizin, Pädiatrische Ernährungsmedizin) beteiligt.
Publikation:
Heekyoung Lee, Kun Qian, Christine von Toerne, Lena Hoerburger, Melina Claussnitzer, Christoph Hoffmann, Viktoria Glunk, Simone Wahl, Michaela Breier, Franziska Eck, Leili Jafari, Sophie Molnos, Harald Grallert, Ingrid Dahlman, Peter Arner, Cornelia Brunner, Hans Hauner, Stefanie M Hauck, Helmut Laumen:
Allele-specific quantitative proteomics unravels molecular mechanisms modulated by cis-regulatory PPARG locus variation, Nucleic Acids Research, 2/2017. DOI: 10.1093/nar/gkx105
Kontakt:
Technische Universität München
Pädiatrische Ernährungsmedizin
Dr. Helmut Laumen
Phone: +49-8161-71-2467
E-Mail: helmut.laumen@tum.de
Helmholtz Zentrum München
Research Unit Protein Science
Dr. Stefanie Hauck
Ph.: +49-89-3187-3941
E-Mail: hauck@helmholtz-muenchen.de
Technische Universität München
Corporate Communications Center
- Sabine Letz
- letz@zv.tum.de
- presse@tum.de
- Teamwebsite