Anhängen von Zuckern verändert die Wirkung von Immunglobulin G-Antikörpern
Antikörper-Bauplan aus dem Rechner
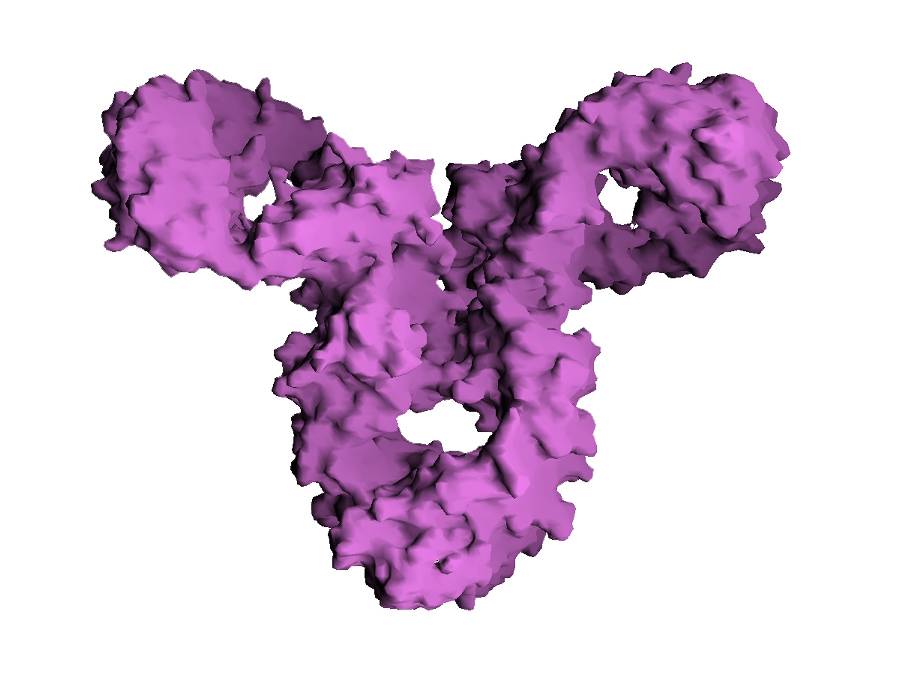
Immunglobulin G-Antikörper, kurz IgG-Antikörper, sind die häufigsten Antikörper im Blut des Menschen und wirken vor allem gegen Viren und Bakterien. Die Antikörper werden durch sogenannte Plasmazellen nach Kontakt mit einem entsprechenden Antigen gebildet.
„Die Antikörper haben die typische Form eines Y und bestehen zu einem Großteil aus Protein“, erklärt Elisa Benedetti, Doktorandin am Institute of Computational Biology (ICB) des Helmholtz Zentrums München. „Allerdings hängt die Zelle bei der Fertigung noch verschiedene Zucker an, und wie das vor sich geht, ist bisher kaum verstanden.“
Je nachdem, welche Zucker angehängt werden, kann der Antikörper sehr unterschiedliche Funktionen ausüben. Während der eine Zucker für eine heftige Entzündungsreaktion beim Kontakt mit dem Antigen sorgt, unterbindet ein anderer die Immunreaktion.
„Kommt es bei der Glykosylierung der Antikörper zu Störungen, kann das daher schwerwiegende Folgen haben“, erklärt Benedetti. So seien bereits mehrere Krankheiten wie Diabetes, Krebs oder Autoimmunerkrankungen, wie beispielsweise Lupus, mit falsch angehängten Zuckern in Verbindung gebracht worden.
Biochemische Aufgabe im Rechner gelöst
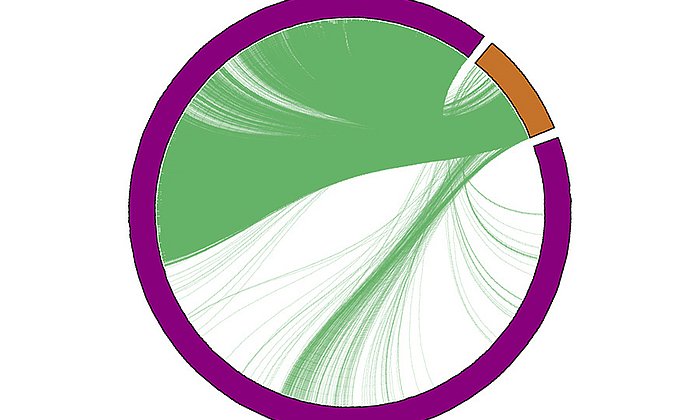
„Die Schwierigkeit bei der Ermittlung der Baupläne liegt unter anderem darin, dass hier mehrere Enzyme beteiligt sind, deren Zusammenwirken sehr komplex ist“, erklärt Dr. Jan Krumsiek, Nachwuchsgruppenleiter am ICB und Junior Fellow der TU München. Um diese biochemisch schwer lösbare Aufgabe anzugehen, holten sich die Bioinformatiker das Problem in die digitale Welt.
Dazu zogen sie Daten der kroatischen „10001 Dalmatiner Biobank“ heran. Darin untersuchten sie zunächst die Blutproben von knapp 700 Probandinnen und Probanden im Alter zwischen 18 und 88 Jahren hinsichtlich der Zucker an den IgG-Antikörpern. Anhand der bisher bekannten Glykosylierungsschritte ermittelten sie, inwiefern die digital erfassbaren Ähnlichkeiten den fortlaufenden enzymatischen Prozess abbilden können.
Vorhersage weiterer Schritte im Reaktionsweg
In der Tat rekonstruierte der Algorithmus anhand der Daten den bisher bekannten Bauplan – und entwickelte ihn weiter. „Wir konnten also neue Schritte vorhersagen, wie die Zuckerreste an den Antikörper gehängt werden müssten“, erklärt Krumsiek. „Anhand drei weiterer Kohorten mit insgesamt über 2.500 Proben konnten wir dieses Modell in der Theorie untermauern.“
Anschließend bestätigten die Forschenden die vorhergesagten Schritte durch verschiedene Methoden: zum einen anhand einer genomweiten Assoziationsstudie mit rund 1.900 Proben einer Studie der Kooperative Gesundheitsforschung in der Region Augsburg (KORA), zum anderen im Rahmen von drei Versuchsreihen im Labor (in vitro).
„Wir konnten zeigen, dass mindestens eine der vorhergesagten Reaktionen im Reagenzglas ablaufen kann. Zudem haben wir in Zellkultur bewiesen, dass bestimmte Enzyme, deren Zusammenwirken unser Modell vorhergesagt hatte, tatsächlich in der Zelle kolokalisiert, also räumlich eng beieinander angesiedelt, sind“, so Krumsiek. „Unsere Studie zeigt, wie sich Informationstechnologien und klassische Nasschemie unterstützen und bereichern können.“
Publikation:
Benedetti, E. et al. (2017): Network inference from glycoproteomics data reveals new reactions in the IgG glycosylation pathway. Nature Communications, DOI: 10.1038/s41467-017-01525-0
Technische Universität München
Corporate Communications Center
- Helge Siemens (HMGU) / Andreas Battenberg (TUM)
- battenberg@zv.tum.de
- presse@tum.de
- Teamwebsite