Quallen-Proteine helfen bei der Erforschung von Herzrhythmusstörungen
Leuchtende Herzzellen
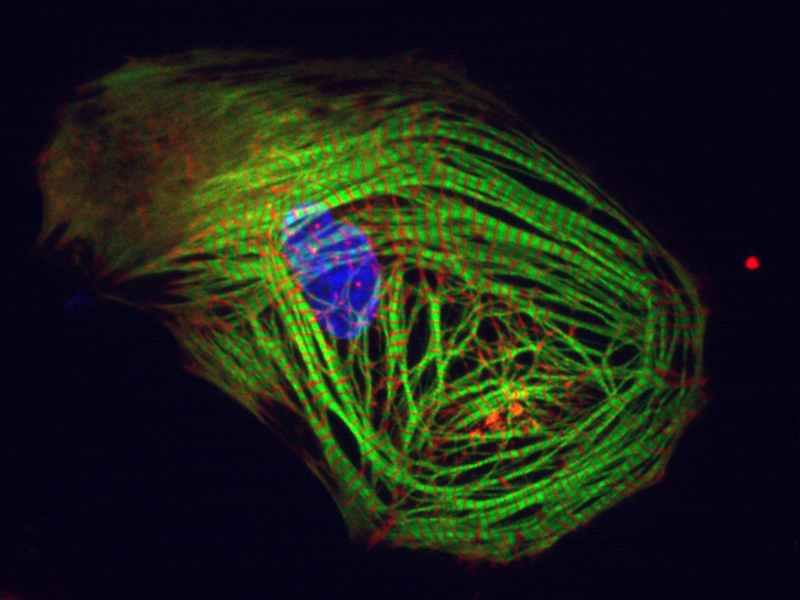
Seit etwa zehn Jahren ist es möglich, aus sogenannten induzierten pluripotenten Stammzellen im Labor Herzzellen herzustellen. Diese Stammzellen werden beispielsweise aus weißen Blutkörperchen gewonnen und können im Labor unbegrenzt vermehrt und zu allen möglichen Körperzellen weitergezüchtet werden. Auf diese Weise hergestellte Herzzellen machen es zum Beispiel möglich, Herzrhythmusstörungen intensiver zu untersuchen, als es bisher möglich war. Tierversuche sind für diese Aufgabe nur sehr bedingt geeignet, und Gewebeproben kann man aus den Herzen von Patientinnen und Patienten auch nicht ohne weiteres entnehmen. In den gezüchteten Herzzellen dagegen lassen sich Erkrankungen sozusagen im Miniaturformat untersuchen.
„Unsere Entwicklung löst gleich mehrere Probleme, die die Arbeit mit solchen Zellmodellen bisher erschwert haben“, sagt Dr. Daniel Sinnecker, Kardiologe am Klinikum rechts der Isar der TUM. Auch bei den Laborzellen stellt sich die Frage danach, wie man ihre elektrische Aktivität am besten messen kann. Bisher wurden für diese Aufgabe meist Mikroelektroden genutzt, mit denen elektrische Signale direkt von den Zellen abgeleitet werden. Das Problem: Diese Prozedur ist sehr aufwendig und erlaubt es daher nur eine sehr kleine Zahl von Herzzellen zu untersuchen.
Unterschiede zwischen Zelltypen
Hinzu kommt, dass Herzzelle nicht gleich Herzzelle ist. Grundsätzlich sind alle Herzzellen in der Lage, sich im Takt selbständig zusammenzuziehen und elektrische Signale an benachbarte Zellen weiterzuleiten. Die Zellen, die die verschiedenen Strukturen des Herzens bilden – beispielsweise die Vorhöfe, die Herzkammern oder den Sinusknoten, den „Taktgeber“ – unterscheiden sich zum Beispiel deutlich in ihren „Aktionspotentialen“. Das sind die Schwankungen in der elektrischen Spannung zwischen Zellinnerem und Zelläußerem, die als elektrisches Signal den Erregungsablauf im Herzen steuern und dafür verantwortlich sind, dass es sich zusammenzieht.
Dieser Unterschied macht sich bei der Untersuchung von Rhythmusstörungen bemerkbar, die auf Fehlfunktionen bestimmter Areale des Herzmuskels beruhen: Züchtet man Herzzellen aus Stammzellen, lässt sich bisher nur unzureichend beeinflussen, ob Herzkammerzellen, Herzvorhof- oder Sinusknotenzellen entstehen. Um welche Sorte es sich handelt, muss man erst mühsam bei jeder einzelnen Zelle feststellen, um eine bestimmte Störung sinnvoll zu untersuchen.
Biologische Sensoren statt Mikroelektroden
Daniel Sinnecker und sein Team beschreiben im „European Heart Journal“ eine mögliche Lösung für diese beiden Probleme. Anstatt den Zellen mit Mikroelektroden zu Leibe zu rücken, versehen die Wissenschaftlerinnen und Wissenschaftler sie mit biologischen Sensoren. Diese sind aus fluoreszierenden, also leuchtenden, Proteinen aus Tiefseequallen aufgebaut. Die DNA, die den „Bauplan“ dieser Sensorproteine enthält, wird in die Herzzellen eingeschleust, woraufhin diese selbst die Sensorproteine herstellen. Werden die so veränderten Herzzellen mit Licht in einer bestimmten Wellenlänge angeregt, leuchten sie in einer anderen Wellenlänge zurück. Die genaue Farbe des zurückgestrahlten Lichts hängt dabei von der Spannungsdifferenz zwischen Zellinnerem und Zelläußerem ab. Mit einer speziellen Kamera kann man deswegen das Aktionspotential der einzelnen Zellen aufzeichnen und messen.
Die Besonderheit der neuen Methode liegt darin, dass die eingeschleuste DNA mit bestimmten Erkennungssequenzen, sogenannten Promotoren, versehen werden kann. Diese sorgen dafür, dass das Sensorprotein nur in bestimmten Typen von Herzmuskelzellen hergestellt wird. So kann man je nach Bedarf gezielt nur die elektrischen Signale aus Vorhofzellen, aus Herzkammer- oder aus Sinusknotenzellen erfassen.
Neue Möglichkeiten für Medikamenten-Tests
Im Gegensatz zu der umständlichen Mikroelektroden-Technik ist diese Methode deutlich leistungsfähiger. „Schon jetzt können wir hunderte Zellen an einem Tag untersuchen statt nur einer Handvoll", sagt Zhifen Chen, Erstautorin der Studie. „Dieser Prozess ließe sich prinzipiell automatisieren und hochskalieren, so dass Tausende Zellen zugleich untersucht werden könnten.“
„In Zukunft könnte man unsere Methode nicht nur anwenden, um Erkrankungen im Labor zu modellieren“, sagt Daniel Sinnecker. „Dadurch, dass wir Zellen in großer Zahl untersuchen können, ließe sich die Methode auch für groß angelegte Medikamententests nutzen, in denen zum Beispiel geprüft wird, ob ein Produkt negative Auswirkungen auf den Herzmuskel hat.“ Eine Herausforderung für solche neuartigen Verfahren liegt darin, die Zellen in der dafür benötigten Menge zu züchten. Daniel Sinnecker und sein Team arbeiten derzeit daran, die Empfindlichkeit ihrer Methode zu steigern.
Originalpublikation
Z. Chen, W. Xian, M. Bellin, T. Dorn, Q. Tian, A. Goedel, L. Dreizehnter, C. M. Schneider, D. Ward-van Oostwaard, J. King Man Ng, R. Hinkel, L. S. Pane, C. L. Mummery, P. Lipp, A. Moretti, K.-L. Laugwitz, D. Sinnecker. "Subtype-specific promoter-driven action potential imaging for precise disease modelling and drug testing in hiPSC-derived cardiomyocytes". Eur Heart J (2016). DOI: http://dx.doi.org/10.1093/eurheartj/ehw189
Pressekontakt
Paul Hellmich
Corporate Communications Center
Technische Universität München (TUM)
Tel.: +49 (89) 289-22731
E-Mail: paul.hellmich@tum.de
Technische Universität München
Corporate Communications Center
- Paul Hellmich
- paul.hellmich@tum.de
- presse@tum.de
- Teamwebsite