Röntgenstrukturanalyse gibt detaillierte Einblicke in molekulare Wirkstoff-Fabrik
Wie bauen Bakterien Naturstoffe auf?
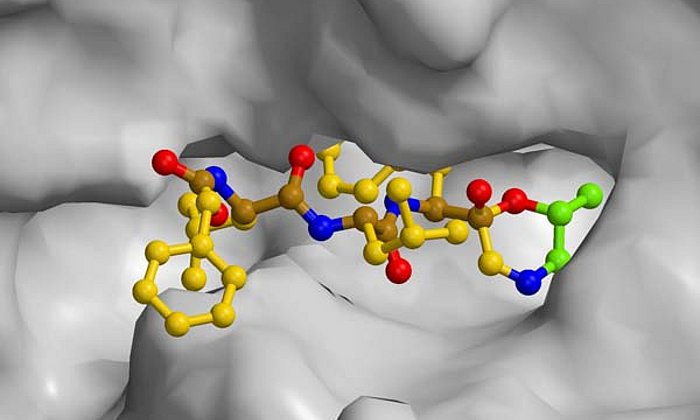
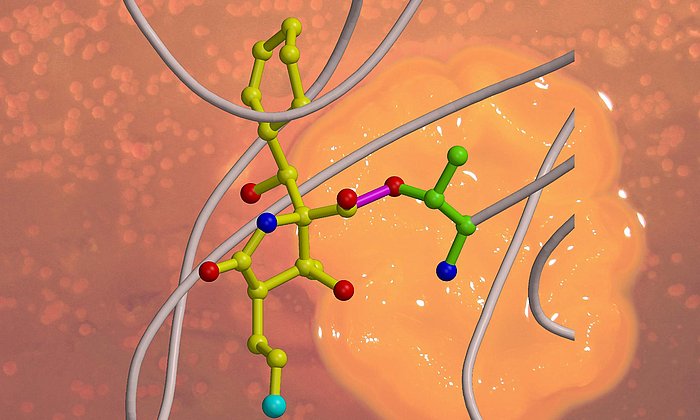
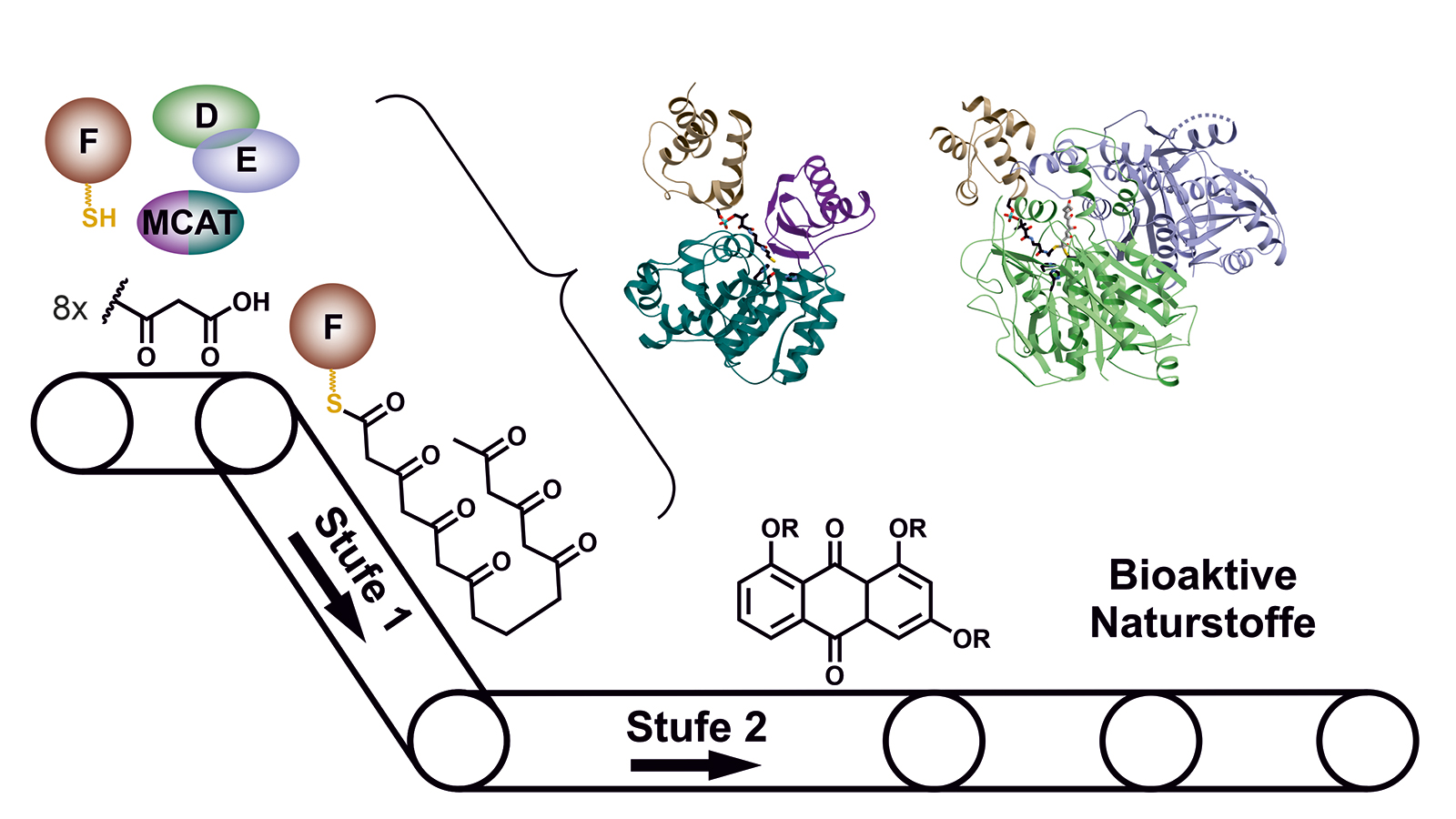
Viele wichtige Medikamente, beispielsweise Antibiotika oder Wirkstoffe gegen Krebs, sind Naturstoffe, die von Mikroorganismen wie zum Beispiel Bakterien oder Pilzen aufgebaut werden. Im Labor können diese Naturstoffe oft gar nicht oder nur mit großem Aufwand hergestellt werden. Ausgangsbasis für eine große Zahl solcher Verbindungen sind Polyketide, Kohlenstoffketten, bei denen jedes zweite Atom eine Doppelbindung zu einem Sauerstoffatom besitzt.
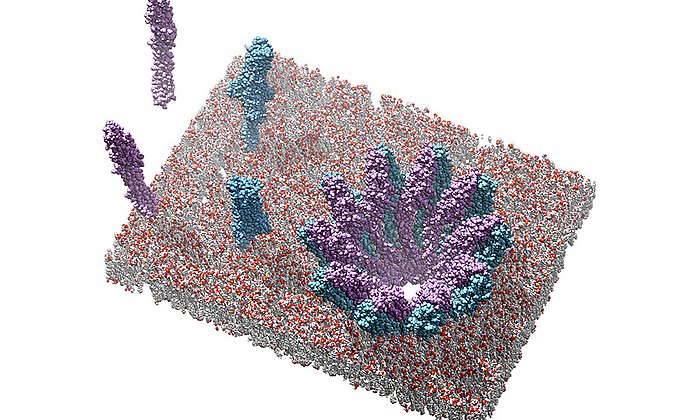
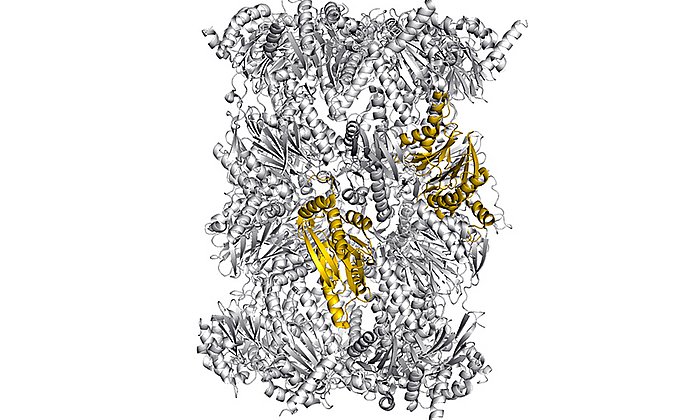
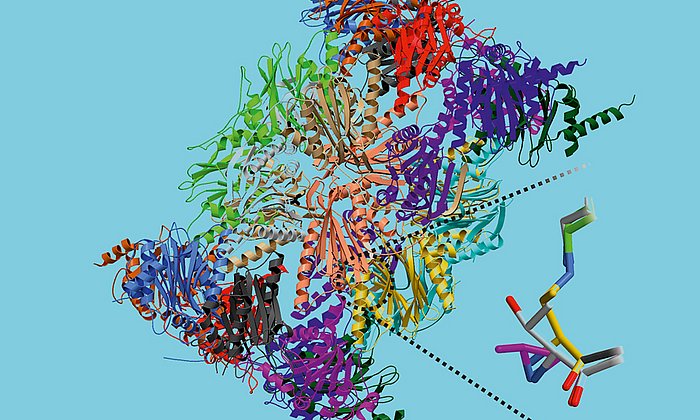
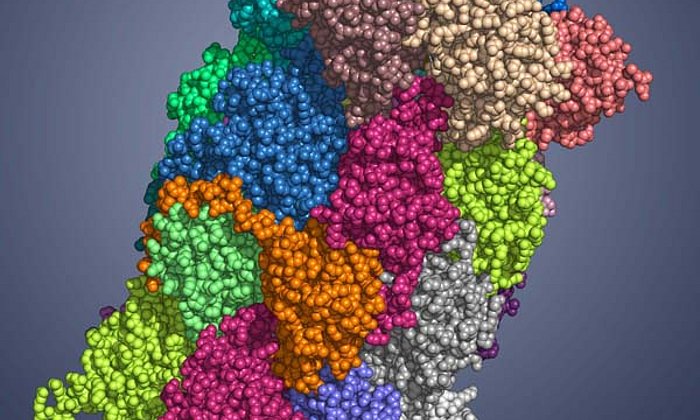
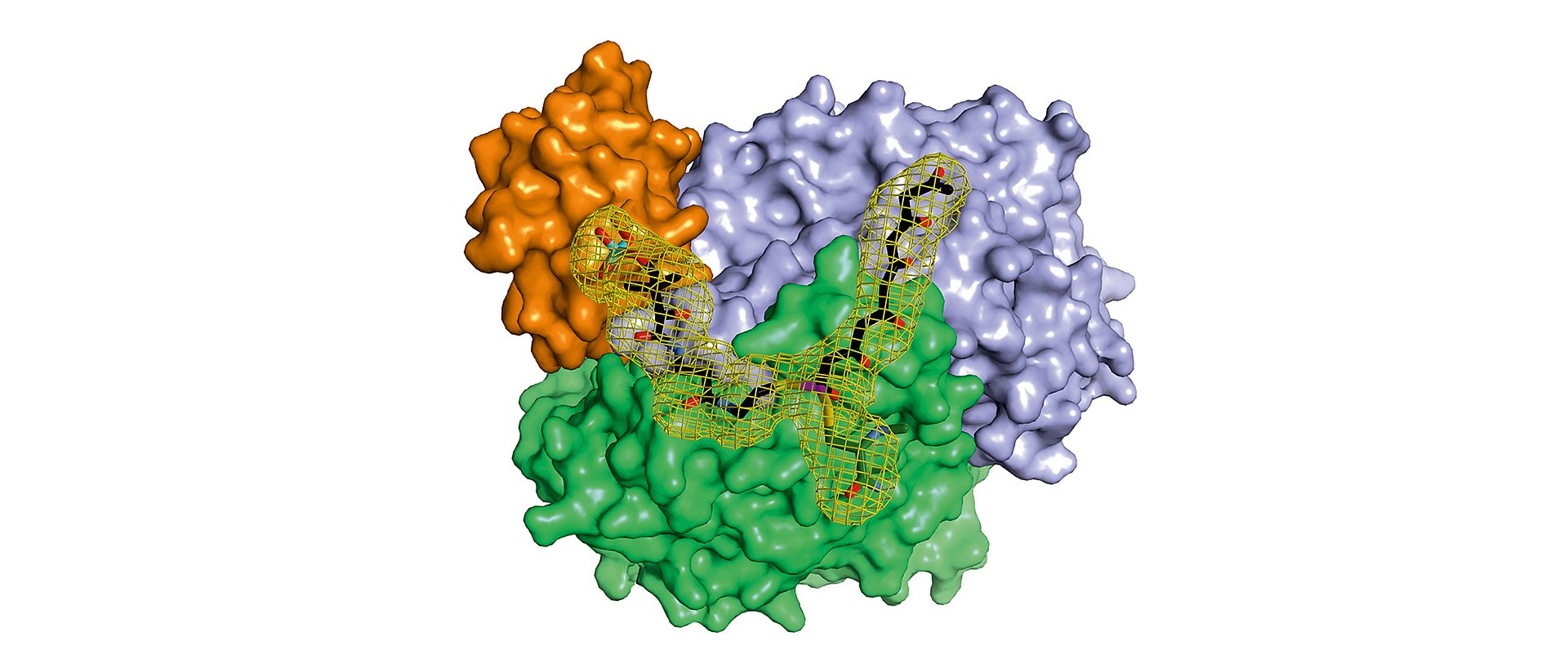
In der Zelle eines Mikroorganismus wie des Bakteriums Photorhabdus luminescens entstehen sie mit Hilfe von Polyketidsynthasen (PKS). Um schrittweise die gewünschten Moleküle aufzubauen, arbeiten bei Typ II PKS-Systemen in der ersten Stufe vier Proteine in wechselnden „Teams“ zusammen.
In einer zweiten Stufe werden diese dann durch weitere Enzyme zum gewünschten Naturstoff modifiziert. Beispiele für so hergestellte bakterielle Naturstoffe sind unter anderem die klinisch genutzten Tetracyclin-Antibiotika oder das Krebsmedikament Doxorubicin.
Interdisziplinäre Zusammenarbeit
Während die modifizierenden Schritte der zweiten Stufe für viele Wirkstoffe gut untersucht sind, gab es bisher kaum Einblicke in die grundsätzliche Arbeitsweise der ersten Stufe dieser molekularen Wirkstoff-Fabriken, bei der das sehr reaktive Polyketid-Zwischenprodukt am Enzymkomplex gebunden und geschützt vorliegt, so dass es nicht spontan reagieren kann.
Diese Lücke schließen nun die im renommierten Fachjournal Nature Chemistry veröffentlichten Ergebnisse der Kooperation zwischen den Arbeitsgruppen von Michael Groll, Professor für Biochemie an der TU München, und Helge Bode, Professor für Molekulare Biotechnologie der Goethe-Universität Frankfurt.
Erkenntnisse inspirieren zu neuen Wirkstoffsynthesen
„Im Rahmen dieser Arbeit konnten wir erstmals Komplexe der verschiedenen Partner-Proteine der Typ II Polyketidsynthase mit Hilfe der Röntgenstrukturanalyse analysieren und so den ganzen katalytischen Zyklus im Detail verstehen“, erläutert Michael Groll.
„Basierend auf diesen Erkenntnissen wird es in Zukunft möglich sein, gezielt in die zentralen biochemischen Prozesse einzugreifen und damit die Grundstrukturen zu verändern, anstatt sich auf die dekorierenden Enzyme zu beschränken“, ergänzt Helge Bode.
Bis verbesserte Antibiotika und andere Medikamente entstehen ist es zwar ein weiter Weg, aber beide Gruppen sind optimistisch, dass nun auch die noch fehlenden Teile der molekularen Fabrik in Struktur und Mechanismus aufgeklärt werden können. „Wir haben bereits vielversprechende Daten von den weiteren Protein-Komplexen“, sagt Maximilian Schmalhofer, der als Doktorand in München an der Studie beteiligt war.
Alois Bräuer, Qiuqin Zhou, Gina L.C. Grammbitter, Maximilian Schmalhofer, Michael Rühl, Ville R.I. Kaila, Helge B. Bode und Michael Groll:
Structural snapshots of the minimal PKS system responsible for octaketide biosynthesis
Nature Chemistry 06.07.2020 – DOI: 10.1038/s41557-020-0491-7
Die Arbeiten wurden gefördert mit Mitteln der Deutschen Forschungsgemeinschaft (DFG) im Rahmen des SPP 1617, des SFB 1035 sowie des Exzellenzclusters Center for Integrated Protein Science München (CIPSM) und des LOEWE Schwerpunkts MegaSyn des Landes Hessen. Röntgenstrukturdaten wurden am Paul Scherrer Institut in Villigen (Schweiz) gemessen. Die Swedish National Infrastructure for Computing stellte Rechenzeit für die theoretische Modellierung zur Verfügung.
Technische Universität München
Corporate Communications Center
- Andreas Battenberg
- battenberg@zv.tum.de
- presse@tum.de
- Teamwebsite
Kontakte zum Artikel:
Prof. Dr. Michael Groll
Professur für Biochemie
Technische Universität München
Lichtenbergstr. 4, 85748 Garching
Tel.: +49 89 289 13360 – E-Mail: michael.groll@tum.de