Synthetische Rotationsmotoren auf der Nanoskala verrichten mechanische Arbeit
Erster elektrischer Nanomotor aus DNA-Material

Ob im Auto, der Bohrmaschine oder der automatischen Kaffeemühle – Motoren verrichten in unserem Alltag Arbeit, um verschiedenste Aufgaben zu bewältigen. Auf deutlich kleinerer Ebene übernehmen natürliche molekulare Motoren lebenswichtige Aufgaben in unserem Körper. So produziert beispielsweise die sogenannte ATP-Synthase das Molekül Adenosintriphosphat (ATP), das unser Körper zum kurzfristigen Speichern und Übertragen von Energie verwendet.
Während natürliche molekulare Motoren essentiell sind, war es bislang eine Herausforderung, Motoren in dieser Größenordnung mit annähernd ähnlichen mechanischen Eigenschaften wie beispielsweise die der ATP-Synthase nachzubauen. Ein Forschungsteam hat nun einen funktionierenden molekularen Rotationsmotor in Nanogröße mit der Methode des DNA-Origami konstruiert. Maßgeblich beteiligt waren die Arbeitsgruppen um Hendrik Dietz, Professor für Biomolekulare Nanotechnologie an der TUM, Friedrich Simmel, Professor für Physik Synthetischer Biosysteme an der TUM sowie Ramin Golestanian, Direktor am Max-Planck-Institut für Dynamik und Selbstorganisation.
Nanomotor setzt sich selbst zusammen
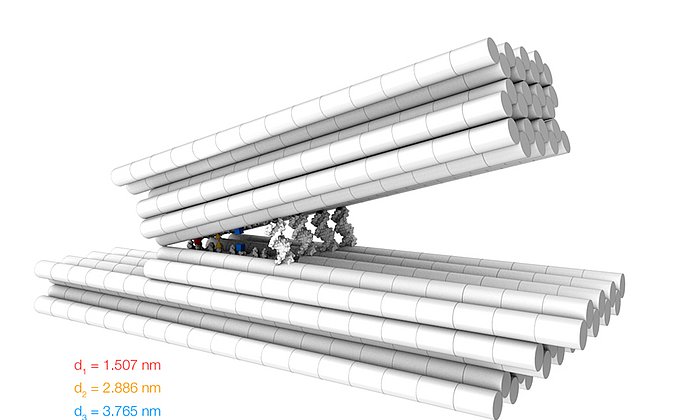
Der neuartige molekulare Motor besteht aus DNA, also Erbgut-Material. Zum Zusammensetzen des Motors aus DNA-Molekülen verwendeten die Forschenden die Methode des DNA-Origami, die 2006 von Paul Rothemund erfunden wurde und in der Folge auch an der TUM in den Arbeitsgruppen maßgeblich weiterentwickelt wurde. Mehrere lange DNA-Einzelstränge dienen dabei als Gerüst. An diesen lagern sich weitere DNA-Stränge als Gegenstücke an. Dabei werden die DNA-Sequenzen so gewählt, dass durch die entsprechenden Anlagerungen und Faltungen die gewünschten Strukturen entstehen.
„Wir arbeiten bereits seit vielen Jahren mit dieser Fabrikationsmethode und können inzwischen sehr präzise und komplexe Objekte entwickeln, wie zum Beispiel molekulare Schalter oder Hohlkörper, die Viren einfangen können. Gibt man die DNA-Stränge mit den entsprechenden Sequenzen in Lösung, setzen sich die Objekte von selbst zusammen“, sagt Dietz.
Der neue Nanomotor aus DNA-Material besteht aus drei Komponenten: Sockel, Plattform und Rotorarm. Der Sockel ist etwa 40 Nanometer hoch und über chemische Verbindungen auf einer Glasplatte in Lösung verankert. Auf dem Sockel ist ein bis zu 500 Nanometer langer Rotorarm drehbar gelagert. Entscheidend für das Funktionsprinzip des Motors ist ein weiteres Bauteil: eine Plattform, die zwischen Sockel und Rotorarm liegt. Diese enthält Hindernisse, die die Bewegung des Rotorarms beeinflussen. Um die Hindernisse zu passieren und sich zu drehen, muss sich der Rotorarm ein wenig nach oben verbiegen, ähnlich wie bei einer Ratsche.
Gezielte Bewegung durch Wechselspannung
Ohne Energiezufuhr bewegen sich die Rotorarme der Motoren getrieben durch zufällige Kollisionen mit Molekülen aus dem umgebenden Lösungsmittel ungesteuert in die eine oder andere Richtung. Sobald jedoch über zwei Elektroden Wechselspannung angelegt wird, drehen sich die Rotorarme gezielt und kontinuierlich in eine Richtung.
„Der neue Motor hat technisch bisher unerreichte mechanische Fähigkeiten: Er kann Drehmomente im Bereich von 10 Piconewton-mal-Nanometer erzielen. Und er kann mehr Energie pro Sekunde erzeugen, als bei der Spaltung von zwei ATP-Molekülen freigesetzt wird“, erklärt Ramin Golestanian, der unter anderem verantwortlich für die theoretische Beschreibung des Motormechanismus zeichnet.
Die gerichtete Bewegung der Motoren entsteht durch eine Überlagerung der fluktuierenden elektrischen Kräfte mit den Kräften, die der Rotorarm aufgrund der Ratschenhindernisse erfährt. Der zugrundeliegende Mechanismus entspricht dabei einer sogenannten „Flashing Brown’schen Ratsche“. Die Forschenden können Geschwindigkeit und Richtung der Rotation über die Feldrichtung und auch über die Frequenz und Amplitude der Wechselspannung kontrollieren.
„Der neue Motor könnte zukünftig auch technische Anwendung finden. Wenn wir den Motor entsprechend weiterentwickeln, könnten wir damit zukünftig eventuell chemische Reaktionen nach dem Vorbild der ATP-Synthase antreiben. Dann könnte man beispielsweise Oberflächen dicht mit solchen Motoren beschichten. Dann gibt man Ausgangsstoffe hinzu, legt ein wenig Wechselspannung an und die Motoren produzieren die gewünschte chemische Verbindung“, spekuliert Dietz.
Anna-Katharina Pumm, Wouter Engelen, Enzo Kopperger, Jonas Isensee, Matthias Vogt, Viktorija Kozina, Massimo Kube, Maximilian N. Honemann, Eva Bertosin, Martin Langecker, Ramin Golestanian, Friedrich C. Simmel & Hendrik Dietz. A DNA origami rotary ratchet motor. Nature (2022). DOI: 10.1038/s41586-022-04910-y
https://www.nature.com/articles/s41586-022-04910-y
- Prof. Hendrik Dietz und Prof. Friedrich C. Simmel forschen am Munich Institute of Biomedical Engineering (MIBE). Das MIBE ist ein Integrative Research Institute der Technischen Universität München (TUM), das interdisziplinäre Zusammenarbeit und Synergien zwischen Forschenden aus dem weiten Feld des Biomedical Engineering fördert. Am MIBE entwickeln und verbessern Forschende aus der Medizin, den Naturwissenschaften und Ingenieurwissenschaften gemeinsam Verfahren zur Prävention, Diagnose und Behandlung von Krankheiten. Die Aktivitäten reichen dabei von der Untersuchung grundlegender wissenschaftlicher Prinzipien bis zu deren Anwendung in medizinischen Geräten, Medikamenten oder Computerprogrammen.
- Forschende folgender Universitäten und Institute waren an den Forschungsarbeiten beteiligt: Technische Universität München (TUM), Max-Planck-Institut für Dynamik und Selbstorganisation, University of Oxford.
- Die Arbeit wurde gefördert von: einem European Research Council Consolidator Grant, der Deutschen Forschungsgemeinschaft über Grants aus dem Gottfried Wilhelm Leibniz–Preis-Programm, dem Sonderforschungsbereich SFB863 „Kräfte in Biomolekularen Systemen“, the Netherlands Organization for Scientific Research, der Max Planck School Matter to Life und dem MaxSynBio Consortium, die beide vom Deutschen Bundesministerium für Bildung und Forschung und der Max-Planck-Gesellschaft finanziert werden.
Technische Universität München
Corporate Communications Center
- Carolin Lerch
- presse@tum.de
- presse@tum.de
- Teamwebsite
Kontakte zum Artikel:
Prof. Dr. Hendrik Dietz
Technische Universität München
Lehrstuhl für Biomolekulare Nanotechnologie
Tel: +49 89 289 11615
dietz@tum.de