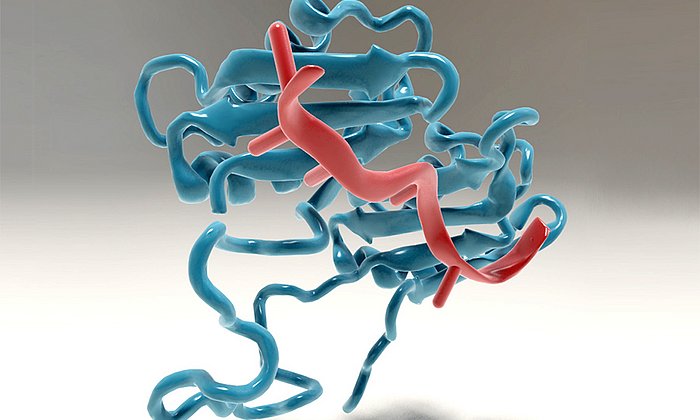
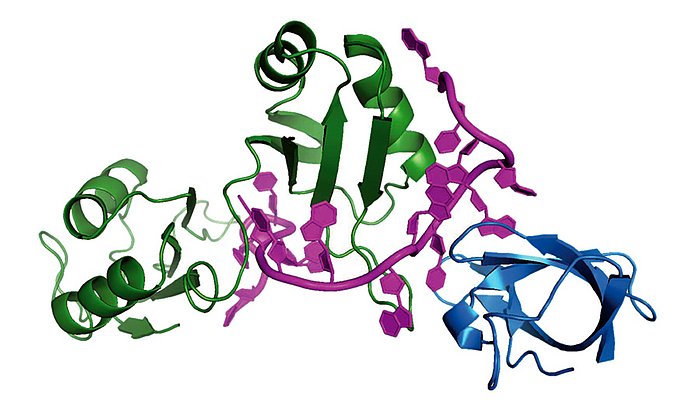
Aufklärung des Reifungsmechanismus einer krebsauslösenden MikroRNA
Neue Einblicke in die Reifung von mikroRNA
MikroRNAs (miRNAs) bilden eine Klasse von Molekülen, die aus kurzen Abfolgen von RNA-Bausteinen bestehen. Sie sind in der Lage, die Herstellung bestimmter Proteine in der Zelle zu verhindern, indem sie die entsprechende Bauplan-RNA abbauen.
Auch krebsauslösende miRNAs, sogenannte oncomiRs, arbeiten nach diesem Prinzip und verhindern vornehmlich die Herstellung von Proteinen, die die Zelle gegen unkontrolliertes Wachstum schützen.
„So führt ein verstärktes Auftreten dieser Moleküle in der Zelle langfristig zur Krebsentstehung“, erklärt Prof. Michael Sattler, Direktor des Instituts für Strukturbiologie am Helmholtz Zentrum München und Lehrstuhlinhaber für Biomolekulare NMR Spektroskopie an der Technischen Universität München (TUM). „Allerdings sind die molekularen Mechanismen, wie manche miRNAs in der Zelle überhaupt hergestellt werden bis heute nicht gut verstanden.“
Reifungsprozess von miRNA
Bevor eine miRNA in der Zelle wirken kann, durchläuft sie mehrere Reifungsschritte und entwickelt sich von einer sogenannten primären pri-miRNA über ein Vorläuferstadium (englisch: precursor, daher pre-miRNA) hin zur reifen miRNA.
Gemeinsam mit Forschenden um Prof. Javier Caceres und Dr. Gracjan Michlewski von der Universität Edinburgh sowie sein Mitarbeiter Hamed Kooshapur (nun National Institutes of Health, USA) untersuchte Sattler in der aktuellen Arbeit die Reifung einer bestimmten pri-miRNA.
„Konkret hatten wir uns auf die Reifung von miRNA-18a konzentriert, die bereits mit Darm-, Brust- und Speiseröhrenkrebs in Verbindung gebracht wurde“, erklärt Michael Sattler. „Um aufzuklären, wie ihre Reifung funktioniert, mussten wir verschiedene Verfahren kombinieren. Dabei kamen sowohl Kernspinresonanz (NMR)-Spektroskopie, Röntgenkristallographie, Kleinwinkel-Röntgenstreuungsanalysen als auch biochemische Experimente zum Einsatz.“
Grundlage für Therapieentwicklung
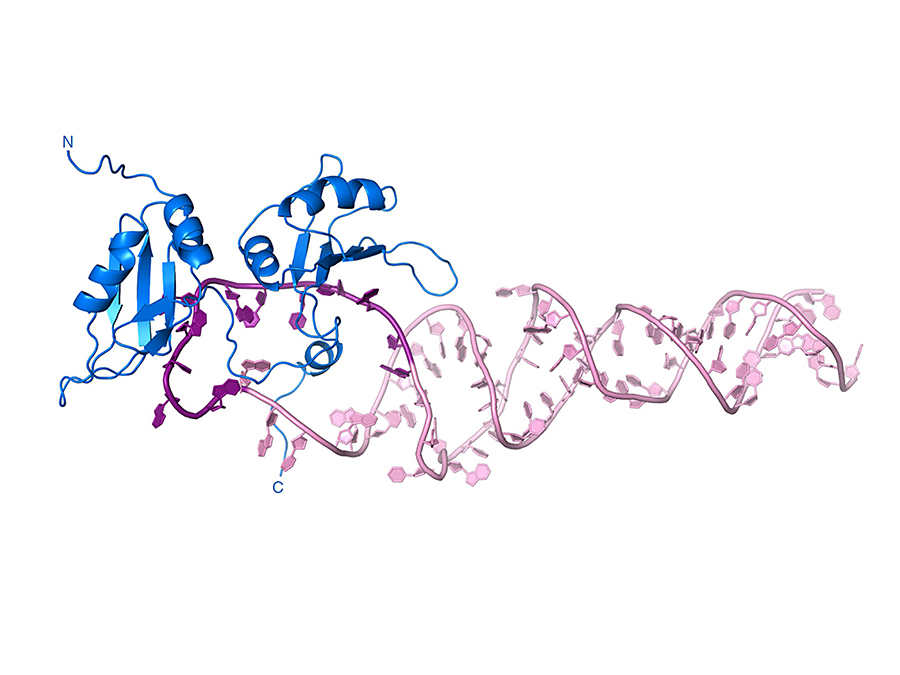
Auf diese Weise konnten die Autoren nachweisen, wie genau ein bestimmtes RNA-Bindeprotein (hnRNP A1) die pri-miRNA-18a erkennt und deren Struktur derart verändert, dass sie sich zur fertigen miRNA-18a weiterentwickeln kann.
Die Forscher gehen davon aus, dass der Mechanismus auch auf andere miRNAs übertragbar ist. „Langfristig hilft uns das Verständnis für die Prozesse dabei, neue Therapieoptionen – beispielsweise gegen Krebs – zu entwickeln“, so Michael Sattler abschließend. „Denn nur wenn wir verstehen, wie die Biologie funktioniert, können wir zielgerichtet eingreifen.“
Publikation:
Hamed Kooshapur, Nila Roy Choudhury, Bernd Simon, Max Mühlbauer, Alexander Jussupow, Noemi Fernandez, Alisha N. Jones, Andre Dallmann, Frank Gabel, Carlo Camilloni, Gracjan Michlewski, Javier F. Caceres and Michael Sattler
Structural basis for terminal loop recognition and stimulation of pri-miRNA-18a processing by hnRNP A1
Nature Communications, (218)9:2479 – DOI: 10.1038/s41467-018-04871-9
Weitere Informationen:
Die Arbeiten wurden gefördert von der Deutschen Forschungsgemeinschaft (SFB 1035, GRK 1721 und Exzellenzcluster Center for Integrated Protein Science Munich), dem Medical Research Council (UK) und dem Wellcome Trust sowie mit Mitteln der Exzellenzinitiative und des Siebten Rahmenprogramms der Europäischen Union über das TUM-Institute for Advanced Study.
SAXS- und SANS-Messungen wurden am DESY (Hamburg) und am Institut Laue-Langevin (Grenoble, Frankreich) durchgeführt, NMR-Messungen am Bayerischen NMR-Zentrum (Garching), Röntgenkristallographie am Helmholtz Zentrum München und weitere SAXS-Messungen am SFB 1035 in der Fakultät für Chemie der Technischen Universität München. Weitere Kooperationspartner waren das Europäische Laboratorium für Molekularbiologie (Heidelberg), die Universität Grenoble Alpes (Frankreich) und die Zhejiang University (China).
Kontakt:
Prof. Dr. Michael Sattler
Technische Universität München
Lehrstuhl für Biomolekulare NMR-Spektroskopie
Lichtenbergstr. 4, 85748 Garching, Germany
Tel.: +49 89 289 13418
michael.sattler@ch.tum.de
Technische Universität München
Corporate Communications Center
- Dr. Andreas Battenberg
- battenberg@zv.tum.de
- presse@tum.de
- Teamwebsite